DNA je nositelem genetické informace v živých organismech. Díky svým chemickým vlastnostem mohou ale molekuly DNA plnit i jiné funkce, např. chovat se jako enzymy a uskutečňovat jednoduché chemické reakce. Edward Curtis, který se se svou skupinou při ÚOCHB zaměřuje na hledání právě takových užitečných molekul DNA, nyní přišel s elegantní metodou, jak toto pátrání v případě některých zajímavých funkcí výrazně urychlit. Svou metodu a výsledky představili vědci v časopise Nucleic Acids Research, s Terezou Streckerovou jako první autorkou.
Funkční molekuly DNA se v současnosti hledají prostřednictvím tzv. in vitro selekce. Tento postup využívá metod umělé evoluce, kdy se vytvoří obrovské množství, až 10 na 15 tedy biliarda či milion miliard náhodných sekvencí DNA, mezi nimiž se hledají molekuly s žádoucími vlastnostmi. Vědci pak tyto obsáhlé soubory různých molekul testují na konkrétní aktivitu. Po každém testovacím kole získají o něco menší soubor, v němž pokračují v hledání. Během klasické in vitro selekce je takový proces nutné mnohokrát opakovat, než se podaří vybrat neboli vyselektovat několik málo sekvencí DNA s požadovanou funkcí. To je úkol podobně náročný jako hledat jedno konkrétní zrnko písku na mořské pláži.
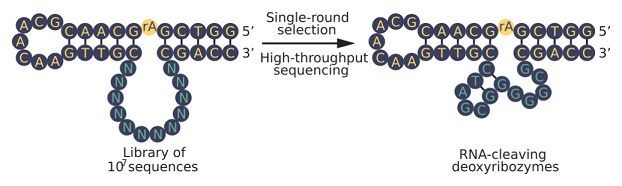
Vědci ze skupiny Edwarda Curtise proto přišli s návrhem metody, která by tento dlouhý a komplikovaný selektivní proces převedla do jediného kroku. Jejím principem je zkrácení úseku DNA, v němž k mutacím dochází, na pouhých 12 či 15 nukleotidů a snížení počtu možných variant DNA řetězců na 10 na 7 nebo 10 na 9. To sice omezuje využití postupu jen na funkce, které nejsou kódovány větším počtem písmen, za to ale umožňuje pracovat se stomilionkrát menšími soubory možných kombinací.
Zdroj: ÚOCHB
Tuto metodu se vědci rozhodli ověřit na modelovém příkladu katalytického štěpení RNA. To je dobře známá a prostudovaná funkce, kterou mají některé sekvence DNA. Nechali si připravit soubor mnoha různých sekvencí, které se lišily pouze v určeném úseku 12 či 15 písmen a současně na vhodném místě obsahovaly jeden ribonukleotid, tedy základní stavební blok RNA. Experiment byl navržený tak, aby sekvence, které by náhodou vykazovaly schopnost katalytického štěpení, mohly snadno reagovat se začleněným ribonukleotidem a řetězec DNA na jeho místě rozštěpily na dvě části.
Po tomto kroku získali soubor mnoha neaktivních dlouhých vláken DNA, mezi nimiž bylo rozptýleno několik kratších, rozštěpených vláken obsahujících molekuly s požadovanými vlastnostmi. Ty pak vědci izolovali ze směsi pomocí elektroforézy využívající toho, že kratší řetězce DNA se pohybují rychleji než delší nerozštěpené molekuly. Následně pomocí sekvenování druhé generace (NGS – next generation sequencing) izolované fragmenty DNA analyzovali a identifikovali mezi nimi nejaktivnější sekvenci, která dokáže štěpit RNA 5000krát rychleji než kontrolní bezenzymové štěpení. Nově objevený deoxyribozym sestávající 12 písmen pojmenovali jednoduše Dvanáctka.
Díky tomuto jednoduchému technickému uspořádání a časové nenáročnosti může nová metoda přinést výrazné zrychlení při odhalování nových krátkých molekul DNA nebo RNA se zajímavými vlastnostmi, např. aptamerů schopných vázat se na konkrétní molekuly a mimikovat tak protilátky. Nový postup by tak mohl být velmi atraktivní pro farmaceutický průmysl.
Streckerová, T.; Kurfürst, J.; Curtis, E. A. Single-round deoxyribozyme discovery. Nucleic Acids Research 2021. https://doi.org/10.1093/nar/gkab504
 Sciencemag.cz
Sciencemag.cz