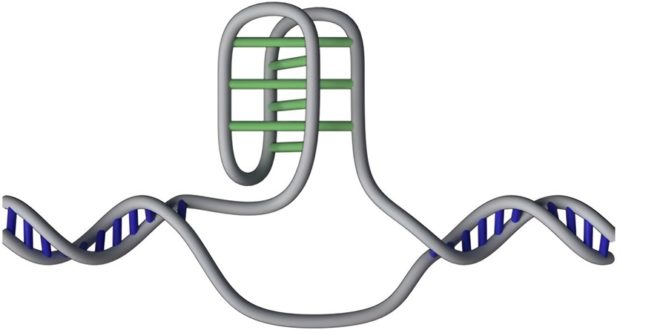
Lidský genom obsahuje neobvyklá místa, zauzlení (knots), kde je DNA čtyřvláknová. Místa vznikají vzájemným párování cytosinů na stejném vláknu. Tyto uzly se označují jako i-motifs. Dosud se o nich skoro nic nevědělo, předpokládalo se ovšem, že budou regulovat čtení „klasických“ genů.
Nyní byla publikována první komplexní mapa těchto struktur DNA, která vrhá světlo na jejich potenciální roli v genové regulaci. Vědci z australského Garvan Institute of Medical Research v roce 2018 poprvé přímo vizualizovali i-motifs uvnitř živých lidských buněk (základní definice i-motif a o objevu v češtině; poněkud překvapivě na webu Ústavu pro jazyk český, totiž ve slovníku neologismů). Nový výzkum vědců z téže instituce stejně jako ten předešlý využívá mapování uzlů pomocí protilátek (imunoglobulinu).
„V této studii jsme zmapovali více než 50 000 i-motifs v lidském genomu, které se vyskytují ve všech třech zkoumaných typech buněk,“ říká hlavní autor studie Daniel Christ. „To je pozoruhodně vysoký počet na strukturu DNA, jejíž samotná existence v buňkách byla kdysi považována za kontroverzní. Naše zjištění potvrzují, že i-motify nejsou jen laboratorní kuriozitou, ale jsou velmi rozšířené – a pravděpodobně hrají klíčovou roli v genomické funkci.“ Ukázalo se, že uzly nejsou v genomu rozmístěny náhodně, ale soustřeďují se v určitých oblastech genomu; těch „funkčních“, ne v oblastech junk DNA. Bývají spojeny s geny, které jsou vysoce aktivní ve specifických obdobích buněčného cyklu. To naznačuje, že hrají dynamickou roli v regulaci genové aktivity.
Uzly byly nalezeny i v promotorech (místech, kde se zahajuje transkripce konkrétního genu) onkogenů včetně známého onkogenu MYC, spojovaného se špatně léčitelnými rakovinami. To naznačuje, že i-moifs by mohly být i cílem léčebných postupů a způsobem, jak regulovat genovou expresi.
Human genomic DNA is widely interspersed with i-motif structures, The EMBO Journal (2024). DOI: 10.1038/s44318-024-00210-5
Zdroj: Garvan Institute of Medical Research / Phys.org
 Sciencemag.cz
Sciencemag.cz